Rafał Ślusarz
Ćwiczenie nr 3.
Słowa kluczowe: pomiary geometrii, wsprowadzenie do współrzędnych wewnętrznych
Przygotowanie
Do dzisiejszego ćwiczenia potrzebne będą następujące pliki:- aspiryna.pdb
- aspiryna.mop
- Molden
- Wyświetlanie
Program Molden uruchamia się poleceniem molden w terminalu. Jako arument wywołania można podać nazwę pliku zawierającego zapisaną strukturę. Programu Molden będziemy używać do wizualizacji struktur zapisanych w formacie współrzędnych wewnętrznych - czego nie potrafi RasMol.Uruchom program Molden wczytując do niego skopiowaną wcześniej strukturę aspiryny (kwasu acetyosalicylowego w formacie "mop") i na tej strukturze sprawdź możliwości wyświetlania tego programu. W szczególności przetestuj, jak działają i co zmieniają w wyświetlaniu funkcje:
- Solid
- StickColor
- Shade
- Perspect.
- Label
- Backbone
Zapoznaj się z operacjami zmiany wyświetlania cząsteczki (Zoom, strzałki kierunków oraz zmiana sposobu rotacji) - Pomiary
Celem dzisiejszego ćwiczenia jest nabranie umiejętności dokonywania pomiarów długości wiązań, odległości między atomami nie związanymi ze sobą kowalencyjnie, oraz wartości kątów walencyjnych i torsyjnych. Jeżeli nie pamiętasz już definicji kątów walencyjnych i torsyjnych, możesz skorzystać z tej strony.Do pomiaru odległości, wartości kąta walencyjnego i torsyjnego w Moldenie służą przyciski oznaczone odpowiednio Distance, Angle i Dihedral. Po wybraniu (kliknięciu) danego przycisku sprawdź w polu Status line, jakiej akcji w danym momencie program wymaga i wykonaj ją.
Zmierz w ten sposób i sprawdź, jak program oznacza zmierzone wielkości, co najmniej jedną odległość między atomami (nie muszą to być atomy połączone ze sobą kowalencyjnie) i po co najmniej jednym kącie walencyjnym i torsyjnym. - Przygotuj grafikę do sprawozdania (w dwóch formatach; spokojnie: dalej jest napisane, jak to zrobić...).
Przygotuj plik o nazwie render.png przedstawiający cząsteczkę aspiryny przygotowany wg wymagań:- reprezentacja space filling
- włączone kolorowanie
- włączone cienie (Shade)
- opisy atomów muszą zawierać symbol pierwiastka i numer kolejny atomu w cząsteczce
Aby z Moldena wyeksportować grafikę, należy użyć przycisku "PostScript" (wybierz opcję PostScript'u kolorowego).
Tak przygotowany plik PS (render.ps) wczytaj do programu graficznego Gimp.
Podczas importu pliku PS do Gimpa:Jeżeli okaże się, że Gimp dzisiaj nie potrafi zaimportować naszego pliku PS, to do zapisania grafiki w formacie PNG można uzyć dwóch innych sposobów:- systemowego PrintScreen (w oknie Moldena: ALT+PrintScreen -> uzyskany plik zapisz w katalogu 'cwiczenie3' pod nazwą render.png). Zapisując plik PNG tym sposobem i tak musisz wyeksportować plik PS z Moldena (będzie on później przesłany na dysk Google, razem ze sprawozdaniem).
- skonwertuj uzyskany plik PS do formatu PDF i dopiero PDF wczytaj do pragramu Gimp. Konwersję możesz wykonac w terminalu wydając polecenie:
ps2pdf render.ps render.pdf
nastepnie wczytaj uzyskany plik PDF do Gimpa i postępuj tak samo, jak gdyby był to pierwotny PS (te same opcje wygładzania i ta sama rozdzielczość).
- włącz najwyższe stopnie wygładzania tekstu i grafiki
- zdefiniuj rozdzielczość importowanego PS na 300 dpi
- Wyświetlanie
- RasMol
- Pomiary
Wczytaj do rasmola odpowiedni plik "pdb" z dzisiejszego zestawu (kopiowany na początku ćwiczeń).
Do pomiaru długości wiązań, wartości kątów walencyjnych i torsyjnych służy menu RasMola Settings - a wnim opcje:
- Pick Distance
- Pick Angle
- Pick Torsion
- Przygotowanie grafiki do sprawozdania
Zlokalizuj i zaznacz na grafice dwa najdłuższe wiązania w cząsteczce aspiryny. Zobrazuj je na grafice pogrubionymi liniami (jedno takie wyróżnione wiązanie znajduje się na grafice w przykładzie w punkcie "Pomiary geometrii", poniżej (uwaga: zaznaczone wiązanie w rzeczywistości wcale nie jest jednym z najdłuższych, ani wypisane wiązania wcale nie muszą występować w tym układzie...)). Wszystkie atomy muszą być podpisane (nazwą atomu i numerem atomu).Wskazówka: Aby wybrać tylko jedno wiązanie i zobrazować je w innym stylu, niż pozostała część cząsteczki, należy wybrać dwa atomy, które je tworzą i zmienić ich tryb wyświetlania na żądany (np. grubszy wireframe). Podstawą powodzenia jest tu wybranie (select) jednocześnie dwóch atomów (RasMol musi zwrócić komentarz: "2 atoms selected!"). Kluczem do sukcesu jest zastosowanie polecenia "select" z dwoma odpowiednimi argumentami rozdzielonymi słowem "or".
Tak przygotowaną strukturę utrwal w postaci zrzutu grafiki (plik graficzny musi się nazywać najdluzsze.png).
- Pomiary
- Pomiary geometrii
Używając dowolnego z poznanych programów do wizualizacji struktur chemicznych przygotuj dokument ODS (arkusz kalkulacyjny OpenOffice), w którym umieścisz tabelę zmierzonych:
- dziesięciu długości wiązań
- dziesięciu wartości kątów walencyjnych
- dziesięciu wartości kątów torsyjnych
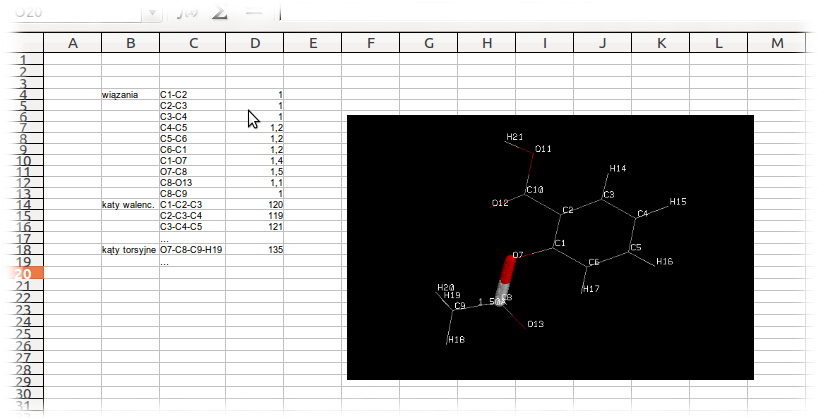
- Sprawozdanie z ćwiczenia.
W sprawozdaniu z ćwiczenia umieść odpowieź na pytania:
- Czy w dowolnym pierścieniu sześcioczłonowym wszystkie wiązania pomiędzy jego węzłami zawsze muszą mieć (i mają) tę samą długość? Dlaczego?
- Czym różnią się pliki tekstowe PDB i MOP? Co zawierają?
- Czy w dowolnym pierścieniu sześcioczłonowym wszystkie wiązania pomiędzy jego węzłami zawsze muszą mieć (i mają) tę samą długość? Dlaczego?
- Udostępnienie do zaliczenia
Na swój dysk Google, do foldera "WDMMcwiczenie3", prześlij pliki:
- render.ps
- render.png
- najdluzsze.png
- pomiary.ods
- sprawozdanie w formacie ODT
- to samo sprawozdanie w formacie PDF
Tak zaopatrzony folder udostępnij prowadzącemu przed końcem zajęć; adres do udostępnień: rafal.slusarz@etoh.chem.univ.gda.pl
WdMM - powrót.